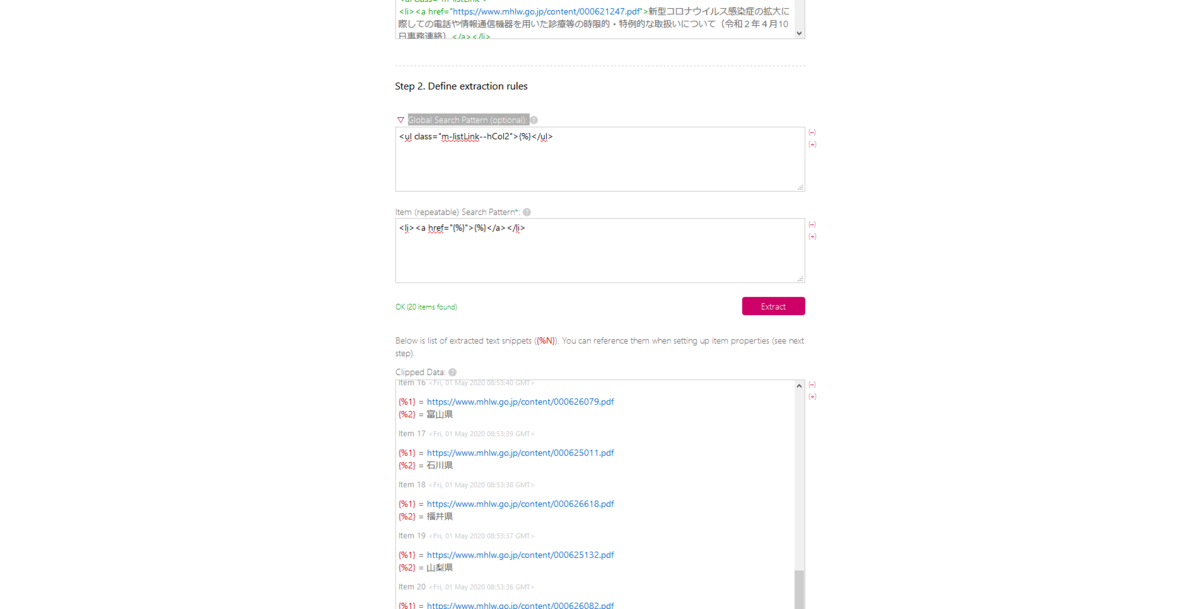
サンプル富山県
github.com
欠損日付を0で補間
df.asfreq("D", fill_value=0)
pandas.pydata.org
import pandas as pd
import matplotlib.pyplot as plt
import japanize_matplotlib
COUNTS_FILE = "toyama_counts.csv"
PATIENTS_FILE = "toyama_patients.csv"
!wget "https://docs.google.com/spreadsheets/d/e/2PACX-1vSJuQThafLPC7OPqUC9TbLV1DmSU0x2Co8VZi2Q2ZZCKLJCTayDl6IoXKyK676mzBgpkoKMgpNK1VML/pub?gid=0&single=true&output=csv" -O $PATIENTS_FILE
!wget "https://docs.google.com/spreadsheets/d/e/2PACX-1vSJuQThafLPC7OPqUC9TbLV1DmSU0x2Co8VZi2Q2ZZCKLJCTayDl6IoXKyK676mzBgpkoKMgpNK1VML/pub?gid=574469870&single=true&output=csv" -O $COUNTS_FILE
df_counts = pd.read_csv(COUNTS_FILE, index_col="年月日", parse_dates=True, dtype={"備考": "object"})
df_counts.head(10)
df_kanja = pd.read_csv(PATIENTS_FILE, index_col="No", dtype={"発症日": "object", "年代": "object", "備考": "object"})
df_kanja.head(10)
df_counts.tail(1)
df_counts["陽性人数"].plot.bar()
df_counts["陽性人数"].cumsum().plot.bar()
df_counts["検査実施人数"].plot.bar()
df_counts["検査実施人数"].cumsum().plot.bar()
df_counts["一般相談件数"].plot.bar()
df_counts["一般相談件数"].cumsum().plot.bar()
df_counts["現在患者数"] = df_counts["陽性人数"] - df_counts["退院者数"] - df_counts["死亡者数"]
df_counts["現在患者数"].plot.bar()
df_counts["現在患者数"].cumsum().plot.bar()
df_counts["退院者数"].plot.bar()
df_counts["退院者数"].cumsum().plot.bar()
age_lists = ["10歳未満", "10代", "20代", "30代", "40代", "50代", "60代", "70代", "80代", "90代以上"]
df_kanja["年代"].value_counts().reindex(age_lists).plot.bar()
df_kanja["居住地"].value_counts().plot.bar()
df_positive = df_counts.loc[:, ["検査実施人数", "陽性人数"]].copy()
df_positive_3d = df_positive.rolling(window=3).mean()
df_positive_3d["陽性率"] = df_positive_3d["陽性人数"] / df_positive_3d["検査実施人数"] * 100
df_positive_3d["陽性率"].plot(ylim=(0, 50))
df_positive_2w = df_positive.rolling(window=7).mean()
df_positive_2w["陽性率"] = df_positive_2w["陽性人数"] / df_positive_2w["検査実施人数"] * 100
df_positive_2w["陽性率"].plot(ylim=(0, 50))
df_positive["陽性率"] = df_positive["陽性人数"] / df_positive["検査実施人数"] * 100
df_positive["陽性率"].plot(ylim=(0, 50))